Najnowsze drzewo życia: wiele nowych grup (wszystkie to bakterie), niektóre tajemnicze i zagadkowe
Wydaje się to dziwaczne, ale tym, co zdarzyło się, jest, że jednokomórkowe stworzenia, jak prymitywne bakterie, rozeszły się na dwie grupy wcześnie w historii życia, a potem jedna z tych grup wyewoluowała w współczesne archeony i współczesne eukarionty. Innymi słowy, jednokomórkowy przodek wszystkich złożonych organizmów był także przodkiem współczesnych archeonów. (Ten jednokomórkowy przodek był prawdopodobnie podobny do żyjących archeonów.) Nie jest trudno to pojąć.
Laura Hug i in. poszli jednak dalej w nowym artykule w „Nature Microbiology” (odnośnik i link poniżej). Poza potwierdzeniem stosunków między trzema wielkimi gałęziami życia, pokazali, że same eubakterie są podzielone na dwie wielkie grupy, które rozeszły się dawno temu. Jedną z nich nazwali Kandydaci na Radiację Typów [Candidate Phyla Radiation] lub CPR. (O pracy Hug i in. napisał także Carl Zimmer w “New York Times” z 11 kwietnia oraz Ed Yong w The Atlantic.)
Wiele archeonów i eubakterii w pracy Hug i in. nie odkryto przez branie znanych organizmów i ustalanie sekwencji ich DNA. Identyfikowano je zamiast tego przez izolowanie fragmentów DNA z różnych środowisk, takich jak solniska, i sekwencjonowanie tych fragmentów raz za razem, aż badacze byli pewni, że każdy genom reprezentuje inny gatunek. Takie organizmy można identyfikować i w zasadzie zrekonstruować w laboratorium, ale ponieważ większości nie można hodować w warunkach laboratoryjnych, nie możemy zobaczyć tych gatunków. Otwiera to zupełnie nowy sposób znajdowania gatunków mimo tego, że ich nie możemy zobaczyć i że możemy wiedzieć bardzo mało o ich naturze.
To, co znaleźli, pokazało możliwość, że istnieją miliony “gatunków” eubakterii i archeonów – pojęcie bakterii staje się nieco mgliste w tych w znacznej mierze aseksualnych grupach – które znajdziemy w przyszłości. Już jest jednak jasne, że Archea i Eubacteria są dominującymi grupami na Ziemi. Nie jest to niespodzianką, ponieważ miały najdłuższy czas na radiację i ich nisze mogą być wszędzie.
Wyniki można podać krótko. Hug i in. skonstruowali drzewo używając znanych sekwencji DNA 2027 gatunków, a następnie zsekwencjonowali genomy 1011 nowych gatunków, odzyskanych przez zbieranie DNA ze środowiska. W sumie mają 3083 gatunki na swoich drzewach. Wybór środowisk dla nowych gatunków jest zabawny:
To badanie włącza 1011 organizmów z linii rodowych, których genomy nie były uprzednio dostępne. Organizmy były obecne w próbkach zebranych z płytkich systemów wodnych, głębokiego, podziemnego miejsca badawczego w Japonii, solnisk na pustyni Atacama, ziemi trawiastej łąki w północnej Kalifornii, bogatego w CO2 systemu gejzerów i z pysków dwóch delfinów.
Pyski delfinów! Ale wyobraźcie sobie jaką liczbę nowych gatunków znaleźliby, gdyby zbierali próbki z jeszcze dziwaczniejszych środowisk, jak dno głębokiego morza lub pyski lam. Pokazuje to, jak mało wiemy o różnorodności jednokomórkowego życia.
Dla skonstruowania drzewa filogenetycznego autorzy użyli genomowego DNA, który koduje prastare białka obecne we wszystkich trzech grupach: białka rybosomalne. Są to białka, które tworzą rybosomy – małe granulki w komórce, w których syntetyzowane są białka przy użyciu matrycowego RNA transkrybowanego z DNA komórki.
Tutaj są te drzewa (szczegóły można zobaczyć w artykule, który jest dostępny za darmo):
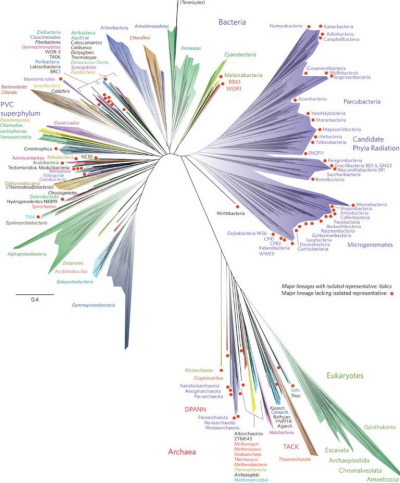
Na drzewie powyżej można zobaczyć trzy wielkie podziały z olbrzymią radiacją bakterii na szczycie (włącznie z fioletową grupą na górze po prawej stronie, niedawno odkryci Kandydaci na Radiację Typów), archeony u dołu po lewej, bliżej spokrewnione z „prawdziwymi” gatunkami komórkowymi niż z bakteriami, i eukarionty, stosunkowo mniejsza grupa na zielono u dołu po prawej. Eukarionty obejmują większość gatunków, które uważamy za interesujące, ale zobaczcie, jak mała jest ta grupa w stosunku do gatunków bakteryjnych. Jest tak, ponieważ przodkowie współczesnej bakterii pojawili się 3,8 miliarda lat temu, ale eukarionty dopiero 1,5 miliarda lat temu. Te pierwsze miały dużo więcej czasu na radiację.
Grupy z czerwonymi punktami to te, którym brak „izolowanych” przedstawicieli: gatunków, które są samotne, są odległymi krewnymi wewnątrz grupy. Nie jestem całkiem pewien, dlaczego autorów to interesowało, ale jestem pewien, że któryś z czytelników mnie poinformuje.
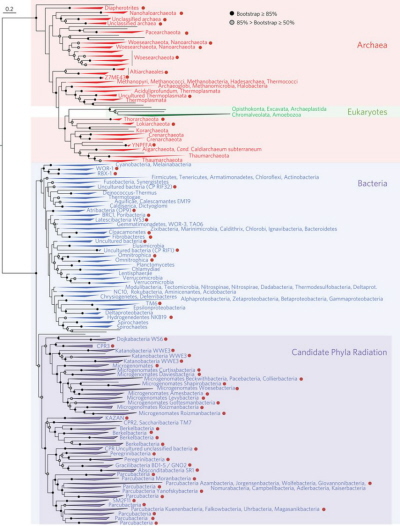
Poniżej pokazuję drzewo Hug i in. oparte na odległości ewolucyjnej (rozchodzenie się sekwencji), co daje jaśniejsze pojęcie o związkach między grupami. Można zobaczyć, że eukarionty są małą grupą wewnątrz archeonów, a więc – przynajmniej dla tych białek – niektóre archeony są bliżej spokrewnione z żyjącymi eukariontami niż z innymi archeonami. Innymi słowy archeony są grupą „parafiletyczną” – taką, która nie obejmuje wszystkich potomków wspólnego przodka (niektórzy potomkowie są eukariontami).
I ostatnia sprawa. Wiele gatunków w nowo odkrytym CPR musi być symbiotyczna (żyjąca w stowarzyszeniu z innymi gatunkami), ponieważ brakuje im genów niezbędnych do niezależnego życia. Jak Hug i in. piszą (moje podkreślenie):
Szczególnie istotni są Kandydaci na Radiację Typów (CPR), podkreśleni fioletowym kolorem w Rys. 1 [pierwszy powyżej]. Informacja dostępna z setek genomów, z metagenomiki i z metod genomiki pojedynczych komórek pokazuje, że wszyscy członkowie mają stosunkowo małe genomy i większość ma nieco (choć niezbyt wysoko) ograniczone możliwości metaboliczne. O wielu wnioskuje się (a co do kilku jest to wykazane) że są symbiontami. Jak dotąd, wszystkim komórkom brakuje pełnych cykli kwasu cytrynowego i łańcuchów oddechowych, a większość ma odgraniczoną lub nie ma żadnej zdolności syntetyzowania nukleotydów i aminokwasów. Pozostaje niejasne, czy ten zredukowany metabolizm jest konsekwencją utraty potencjału w całym nadtypie, czy też są to odziedziczone cechy, które wskazują na wczesną metaboliczną platformę życia. Jeśli jest to odziedziczone, to przyjęcie symbiotycznego stylu życia może być późniejszą innowacją tych organizmów, kiedy już pojawiły się bardziej złożone organizmy.
Nazywają te gatunki “symbiotycznymi”, sugerując, że otrzymują niezbędne aminokwasy i nukleotydy od innych gatunków, ale te symbionty mogą być albo mutualistyczne (oba gatunki odnoszą korzyści), amensalne (gatunek CPR odnosi korzyści, nie ma kosztu dla drugiego gatunku) lub pasożytnicze (gatunek CPR odnosi korzyści kosztem partnera/ów). Zarówno to, jak sama radiacja CPR były w znacznej mierze niespodzianką. Wraz z olbrzymią różnorodnością gatunków znalezionych przez sekwencjonowanie DNA ze środowiska są to dwa wielkie wyniki tego badania.
Niestety, chociaż w zasadzie można zrekonstruować te tajemnicze bakterie z ich genomów – możemy zsyntetyzować genomy CPR i wstrzyknąć je następnie do żywej bakterii, z której usunęliśmy jej DNA – nigdy ich naprawdę nie zobaczymy, bo nie możemy hodować większości z nich w laboratorium. Na razie i dopóki nie rozwiniemy lepszych technik hodowlanych, będziemy żyć w świecie pełnym gatunków, których istnienie możemy rozpoznać, ale których cech nie widzimy.
________
Hug, L. A. et al. 2016. A new view of the tree of life. Nature Microbiol. doi:10.1038/nmicrobiol.2016.48
The newest tree of life: many new groups (all bacteria) some cryptic and enigmatic
Why Evolution Is True, 13 kwietnia 2016
Tłumaczenie: Małgorzata Koraszewska
Profesor na wydziale ekologii i ewolucji University of Chicago, jego książka "Why Evolution is True" (Polskie wydanie: "Ewolucja jest faktem", Prószyński i Ska, 2009r.) została przełożona na kilkanaście języków, a przez Richarda Dawkinsa jest oceniana jako najlepsza książka o ewolucji. Jerry Coyne jest jednym z najlepszych na świecie specjalistów od specjacji, rozdzielania się gatunków. Jest wielkim miłośnikiem kotów i osobistym przyjacielem redaktor naczelnej.