Sposób znajdowania genów choroby
Na drugim krańcu masz to – badanie, w którym przeprowadzono bardzo wiele eksperymentów, żeby zidentyfikować 18 genów leżących u podłoża dziedzicznego kurczowego porażenia kończyn (HSP). Jest to zróżnicowana grupa zaburzeń genetycznych, w której następuje uszkodzenie długich neuronów biegnących między mózgiem a rdzeniem kręgowym, prowadząc do sztywnienia i mimowolnych skurczy mięśni nóg.
Naukowcy już powiązali 22 geny z HSP, ale wyjaśniają one tylko 20 do 30 procent przypadków. “Wiele dzieci z tym zaburzeniem nie może być właściwie zdiagnozowanych i nie ma żadnego dostępnego leczenia” – mówi Joseph Gleeson z University of California, San Diego. “Chcemy dowiedzieć się więcej o przyczynach i mamy nadzieję, że pomoże to w szukaniu jakichś nowych terapii”.
W celu znalezienia większej liczby genów HSP zespół Gleesona nawiązał kontakt z naukowcami w krajach, gdzie HSP występuje częściej i gdzie badania genetyczne są rzadkością: Egiptem, Pakistanem i Iranem. Znaleźli 55 rodzin z tym zaburzeniem i zsekwencjonowali każdy gen 93 osób z tych rodzin. Zidentyfikowali kilka genów, które wydawały się powodować HSP u tych ludzi i wyhodowali zmutowane ryby, żeby sprawdzić, czy pozbycie się tych genów rzeczywiście daje właściwe objawy. Stworzyli sieć, żeby pokazać, co te geny robią i jak wpływają wzajem na siebie. I użyli tej sieci do znalezienia jeszcze większej liczby genów HSP.
Zasięg tej pracy, prowadzonej przez członków zespołu Gaię Novarino, Ali Fenstermaker i Mahę Zaki, jest niewiarygodnie szeroki. “Pracowaliśmy nad tym przez blisko 10 lat – mówi Gleeson. – Po prostu nie chcieliśmy publikować zanim cała praca nie została ukończona. Mamy nadzieję, że ludzie spojrzą na nasz artykuł jak na mapę drogową do badania genetycznie różnorodnych schorzeń”.
18 nowych genów wraz z 22 starymi wyjaśnia około 70 procent przypadków HSP wśród pacjentów badanych przez ten zespół. “To ma olbrzymią wartość, nie tylko dla biologicznego zrozumienia, ale dostarcza to także rodzinom lepszej diagnozy i przyspiesza badania nad możliwymi terapiami dla tych postępujących zaburzeń” – mówi Joris Veltman, genetyk z Radboud University Nijmegen Medical Centre.
Już znalezienie rodzin było wystarczająco trudne. „Nie jest łatwo Amerykaninowi wjechać do Iranu” – mówi Gleeson. Ale było warto, bo w tych częściach świata praktyka małżeństw między krewnymi oznacza, że członkowie rodziny podzielają niezwykle wysoki procent DNA. Ułatwia to znalezienie genów recesywnych, które powodują, że ludzie chorują na HSP tylko, jeśli odziedziczyli dwie kopie.
Zespół zsekwencjonował pełne egzomy – jeden procent ich genomu, który koduje białka - każdego uczestnika. Przez porównanie egzomów członków rodziny z lub bez HSP pokazali, że jedna trzecia przypadków była spowodowana tymi genami, które już uznawano za wmieszane w powodowanie tego zaburzenia. Pozostałe 40 procent mogły być spowodowane przez mutację w 15 innych genach.
Zweryfikowali następnie tę listę przez genetyczne zmanipulowanie ryb z gatunku Danio pręgowany, którym brakowało każdego z tych wytypowanych genów. Żaden z mutantów nie potrafił porządnie pływać. Niektóre miały na przykład ogony na stałe wykrzywione na bok, podobnie do sztywnych kończyn dzieci z HSP. „Uważaliśmy, że musimy to zrobić – mówi Gleeson. – Dla wielu z tych genów mieliśmy tylko po jednej rodzinie z mutacją. Bez eksperymentu z rybami nie czulibyśmy się pewnie twierdząc, że te geny rzeczywiście mają związek z HSP”.
Sekwencjonowanie egzomu szybko staje się pierwszoliniową techniką dla detektywów genów, którzy nie muszą już zawężać poszukiwań do określonych części genomu. Mogą po prostu zsekwencjonować wszystkie geny i zobaczyć, co im wyskoczy. „To badanie wyraźnie przesuwa takie podejście na wyższy poziom, przez zastosowanie go do bardzo dużej kohorty i dokonanie systematycznych badań potwierdzających” – mówi Veltman.
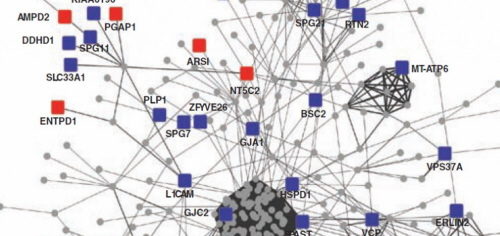
Ale nawet to im nie wystarczyło. „W niektórych chorobach człowiek zostaje z mieszaniną genów i bez żadnej wyraźnej ścieżki przed sobą – mówi Gleeson. – Próbowaliśmy spleść cechy wspólne w naszych genach i zobaczyć, co nam mówią”. Zrobili to przez zmapowanie wszystkich interakcji między genami HSP i białkami, jakie produkują, tworząc splątaną sieć, którą nazwali „HSPome”.
Geny gromadziły się w różnych grupach, zależnie od tego, co robiły. „To było jak podniesienie zasłony – mówi Gleeson. – Mogliśmy zobaczyć, jak wszystkie czynniki, które zostały zidentyfikowane, pasują do siebie”. Jedne były zaangażowane w poprawne składanie białek, inne pomagały w tworzeniu cegiełek DNA, a jeszcze inne pomagały neuronom rosnąć i przesuwać się na właściwe miejsce. Te zgrupowania mówią nam o „punktach wrażliwości molekularnej” w neuronach „od mózgu do kręgosłupa”, które są uszkodzone w HSP, mówi John Fink z University of Michigan.
Zespół rozszerzył następnie sieć, żeby spojrzeć na inne geny, które wchodzą w interakcje z genami zidentyfikowanymi – “przyjaciele przyjaciół”. Przez zeskanowanie tej rozszerzonej listy znaleźli trzy dodatkowe geny HSP, które powodowały chorobę w kolejnych trzech rodzinach. Doprowadziło to do sumy 18 genów.
Sieć nakładała się także w znacznej mierze na inne zestawy genów, które podejrzewano w chorobie Alzheimera, Parkinsona i Lou Gheriga. Sugeruje to, że te różne choroby mózgu mogą mieć jakąś wspólną podstawę i że leki celujące w te zachodzące na siebie geny mogą pomóc w leczeniu kilku chorób.
“To jest ważne, ponieważ wytworzenie leku jest bardzo kosztowne i im większy potencjalny rynek, tym bardziej zainteresowane będą firmy farmaceutyczne do pójścia tą drogą” - mówi Craig Blackstone z National Institutes of Health. Rzeczywiście, przez powiązanie HSP z lepiej badanymi (i lepiej finansowanymi) chorobami może, zdaniem Gleesona, wywołać zainteresowanie tymi często pomijanymi schorzeniami.
“To są fascynujące czasy na badanie nie tylko przyczyn i terapii na HSP, ale także innych chorób neurodegeneracyjnych” – mówi Fink.
Źródło: Novarino, Fenstermaker, Zaki Hofree, Silhavy, Heiberg, Abdellateef, Rosti, Scott, Mansour, Masri, Kayserili, Al-Aama, Abdel-Salam, Karminejad, Kara, Kara, Bozorgmehri, Ben-Omran, Mojahedi, Gamal El Din Mahmoud, Bouslam, Bouhouche, Benomar, Hanein, Raymond, Forlani, Mascaro, Selim, Shehata, Al-Allawi, Bindu, Azam, Gunel, Caglayan, Bilguvar, Tolun, Issa, Schroth, Spencer, Rosti, Akizu, Vaux, Johansen, Koh, Megahed, Durr, Brice, Stevanin, Gabriel, Ideker, and Gleeson. 2013. Exome Sequencing Links Corticospinal Motor Neuron Disease to Common Neurodegenerative Disorders. Science http://dx.doi.org/10.1126/science.1247363
Now this is how you find disease genes
Not Exactly Rocket Science, 30 stycznia 2014
Tłumaczenie M.K.
Ed Yong
Mieszka w Londynie i pracuje w Cancer Research UK. Jego blog „Not Exactly Rocket Science” jest próbą zainteresowania nauką szerszej rzeszy czytelników poprzez unikanie żargonu i przystępną prezentację.
Strona www autora